Bazy sekwencji genomowych w połączeniu z wielkoskalową strukturalną analizą in silico mogą skutecznie prognozować właściwości potencjalnych enzymów o znaczeniu medycznym. Poznańskie Centrum Superkomputerowo-Sieciowe pozwoliło zastosować rozwinięte oprogramowanie do analizy ponad 256 mln białek zawartych w 85 tysięcy genomów bakteryjnych.
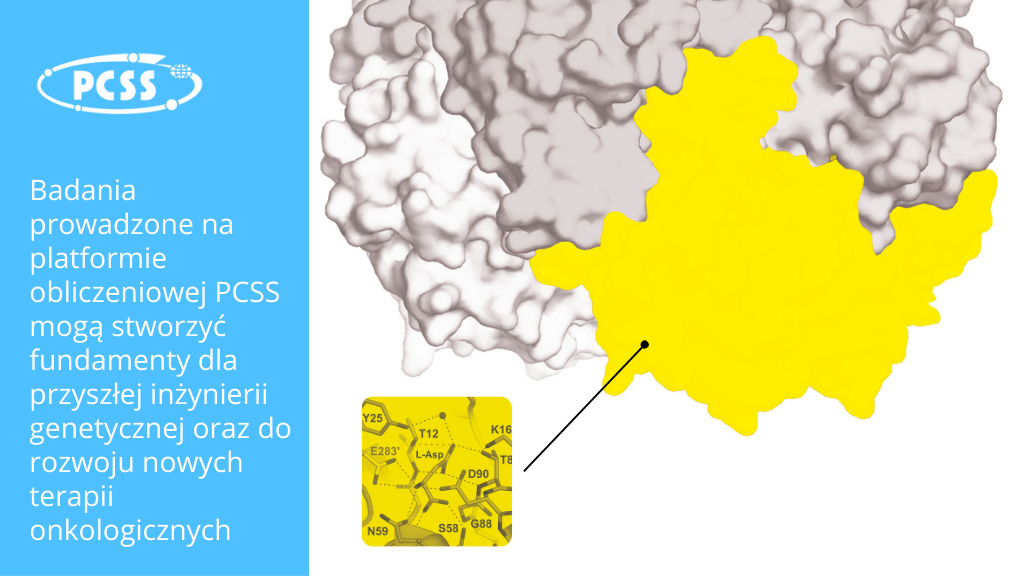
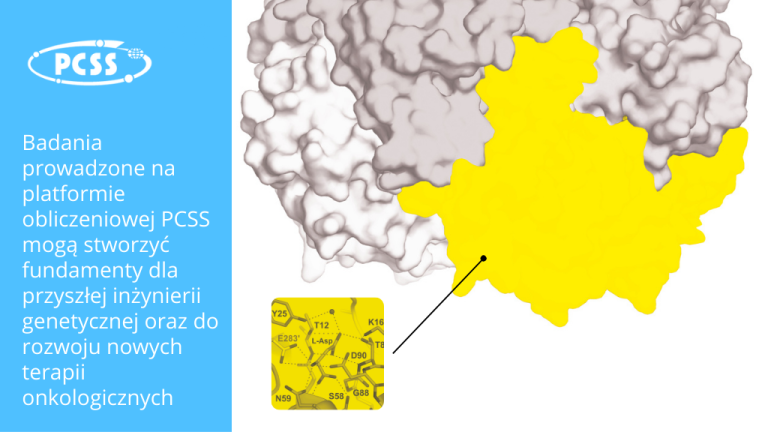
L-asparaginazy to enzymy hydrolizujące L-asparaginę do L-asparaginianu oraz amoniaku. Enzymy te dzielą się na trzy klasy strukturalne, jednak tylko asparaginazy klasy pierwszej, dzięki swojemu silnemu oddziaływaniu z L-asparaginą, są wykorzystywane z powodzeniem w terapii białaczki. Komórki nowotworowe są zależne od zewnętrznego źródła L-asparaginy i wymagają dostaw tego aminokwasu z surowicy krwi do swojego wzrostu i podziału. Efekt terapeutyczny L-asparaginazy polega na obniżeniu poziomu L-asparaginy w krwi pacjenta, prowadząc do zahamowania wzrostu komórek nowotworowych. Obecnie w terapii onkologicznej zatwierdzone są jedynie enzymy z dwóch źródeł organizmów bakteryjnych, z Erwinia chrysanthemi (ErAII) oraz Escherichia coli (EcAII). Ostatnie badania sugerują, że potencjał terapeutyczny L-asparaginaz nie ogranicza się jedynie do białaczek i można je zastosować w leczeniu innych typów nowotworów, np. chłoniaków, glejaków czy nowotworów trzustki. Istnieje zatem duże zainteresowanie projektowaniem nowych terapeutycznych L-asparaginaz. Dodatkową motywacją jest również fakt, że stosowane obecnie terapeutyki (ErAII oraz EcAII) mimo swojej skuteczności wywołują również wiele skutków ubocznych.
W projekcie opracowano i stworzono linię obliczeniową do identyfikacji L-asparaginaz, przewidywania ich struktur przestrzennych i analizowania właściwości, zwłaszcza powinowactwa L-asparaginy, w porównaniu z enzymami stosowanymi obecnie w terapii nowotworowej. Opracowane narzędzie umożliwia identyfikację sekwencji L asparaginaz w genomowych bazach danych, generując trójwymiarowe modele wytypowanych białek, a następnie wykorzystuje techniki dokowania, dynamiki molekularnej i chemii kwantowej do oszacowania względnego powinowactwa tych białek do L-asparaginy. Zaawansowana infrastruktura obliczeniowa Poznańskiego Centrum Superkomputerowo-Sieciowego pozwoliła zastosować rozwinięte oprogramowanie do analizy ponad 256 mln białek zawartych w 85 tys. genomów bakteryjnych. Dzięki tym badaniom udało się zidentyfikować kilkaset enzymów o potencjalnie wyższym powinowactwie do substratu niż obecnie stosowane w terapii. Wybrane enzymy wytypowane na podstawie metod obliczeniowych zostaną wyprodukowane w prokariotycznym systemie ekspresyjnym, oczyszczone za pomocą technik chromatograficznych, a następnie ich powinowactwo do substratu zostanie sprawdzone in vitro. Enzymy o najlepszym powinowactwie do L-asparaginazy i odpowiednich parametrach fizykochemicznych będą testowane na wybranych liniach ludzkich komórek nowotworowych w celu oceny ich zdolności do hamowania proliferacji i indukowania apoptozy w tych komórkach, co będzie pierwszym krokiem do ich wykorzystania w terapii.
dr Maciej Janicki
Pracownia Biotechnologii, Wydział Biologii UAM
dr Andrzej Zieleziński
Zakład Biologii Obliczeniowej, Wydział Biologii UAM
dr hab. Joanna Loch
Zakład Krystalochemii i Krystalofizyki, Wydział Chemii UJ